ENCODE RNA-seq、eCLIP-seq应用 | SNV影响RNA剪切
“手上掌握了许多RNA-蛋白质结合数据,怎么用?”
UCLA的Xinshu (Grace) Xiao, Ph.D.用ENCODE的RNA-seq找出622个GMAS (影响可变剪切的内含子SNV标签),又用ENCODE的eCLIP-seq数据阐释了GMAS的调控机理。


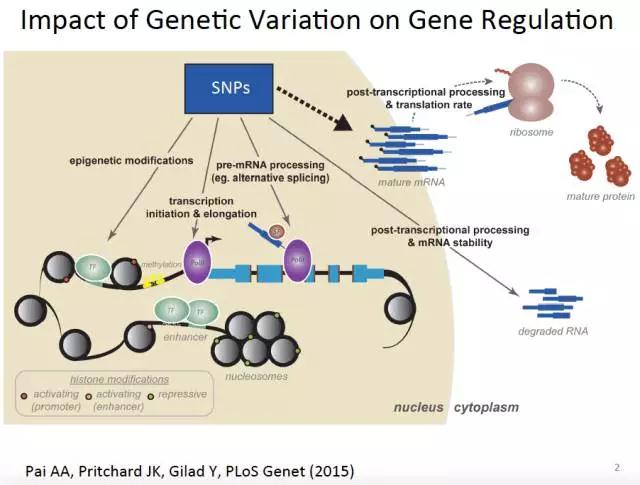
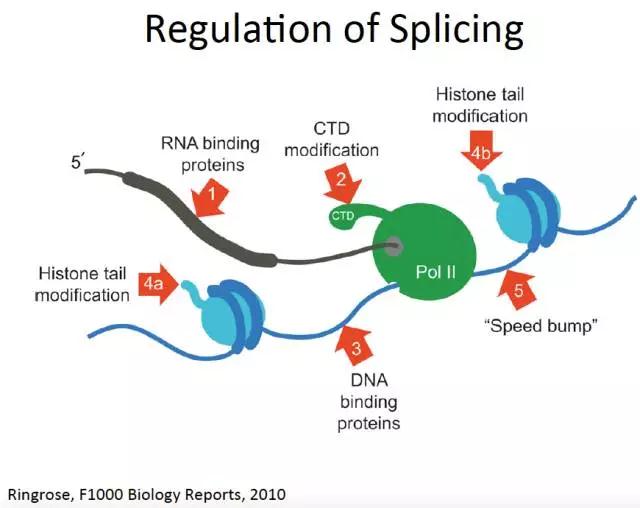
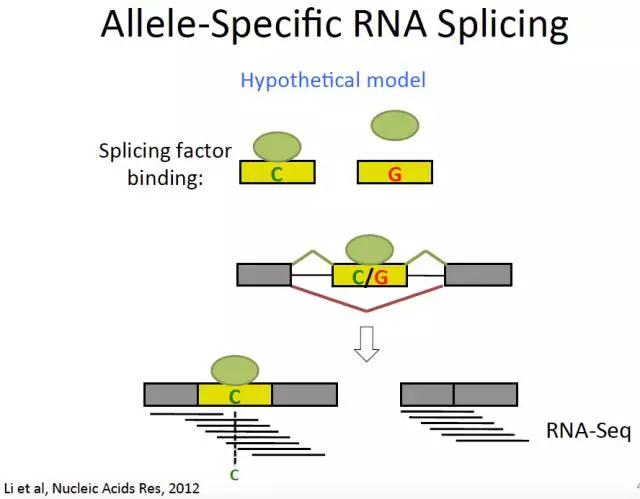
DNA序列的改变可能导致:本来应该结合的剪切因子无法结合
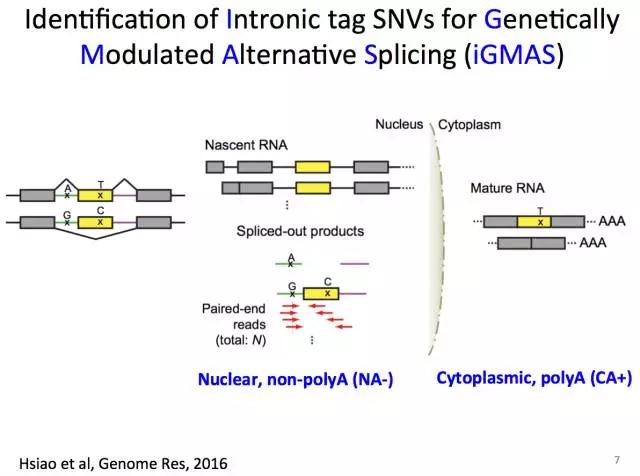
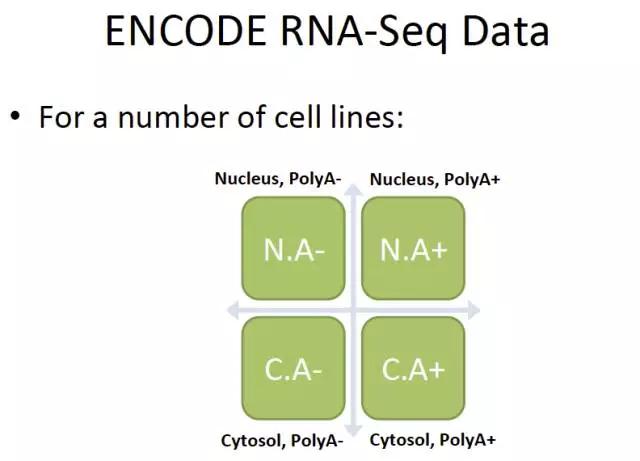
下面是研究RNA剪切加工的理想实验设计:
细胞核里带PolyA的RNA;
细胞质里带PolyA的RNA;
细胞核里不带PolyA的RNA;
细胞质里不带PolyA的RNA;
这些统统分开测
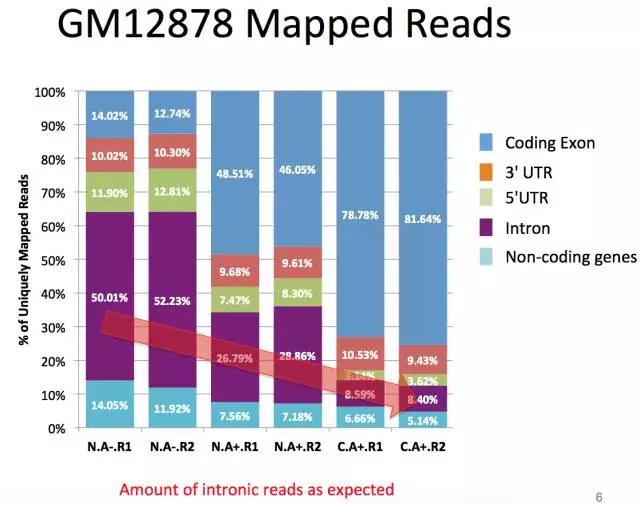
这样测得的RNA-seq数据,能明显看到intron被剪掉的过程。
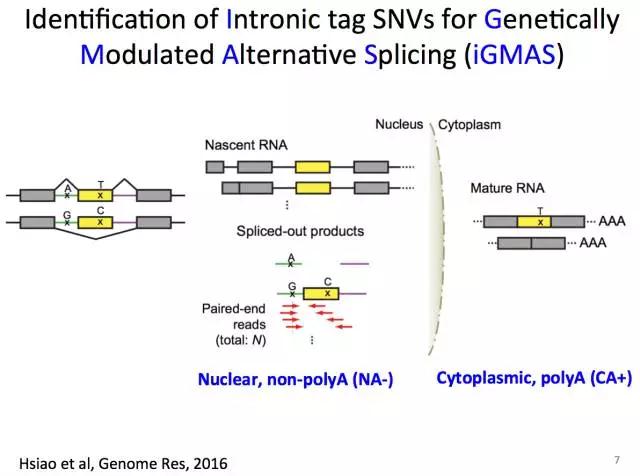
从中鉴定出iGMAS:影响可变剪切的内含子SNV标签
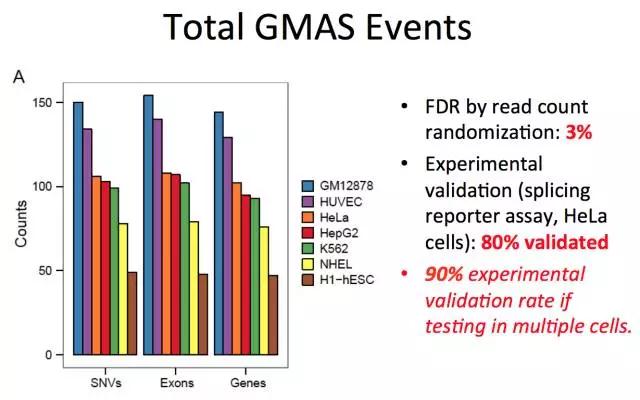
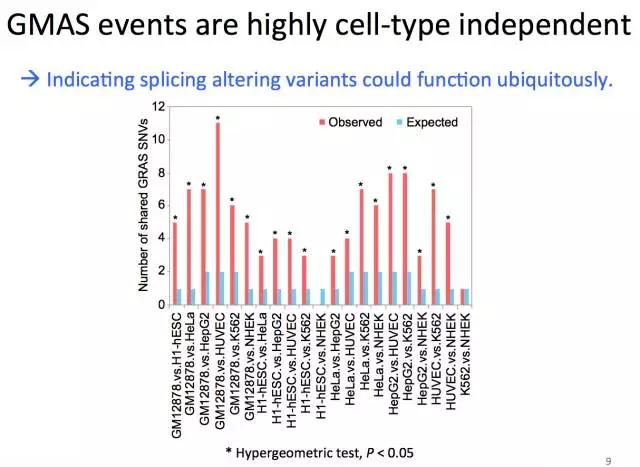
貌似GMAS广泛存在于各种细胞类型,那么这种内含子上的变异有什么影响呢?

先预测,认为以下剪切因子可能结合这些GMAS位点。
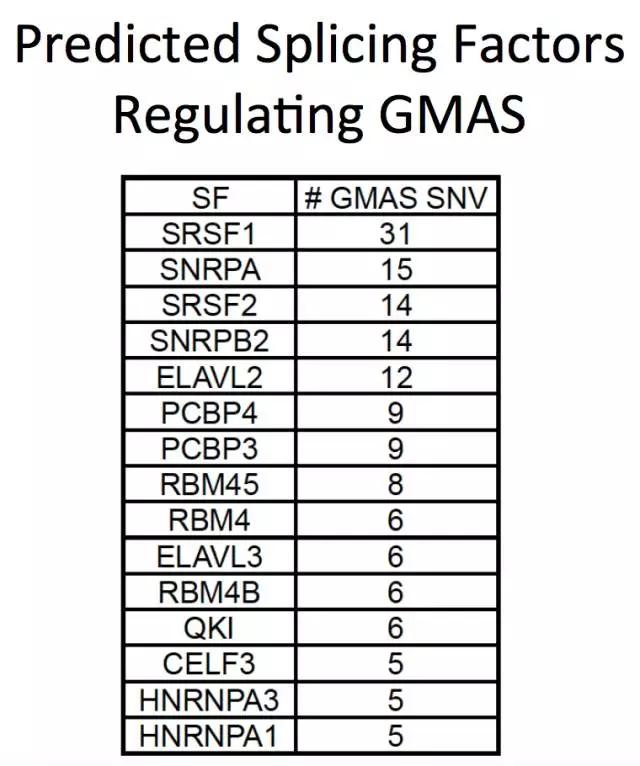
再用ENCODE的eCLIP-seq数据验证:
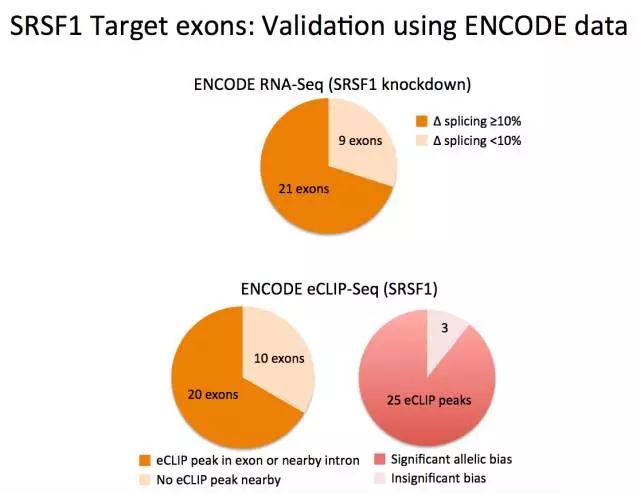
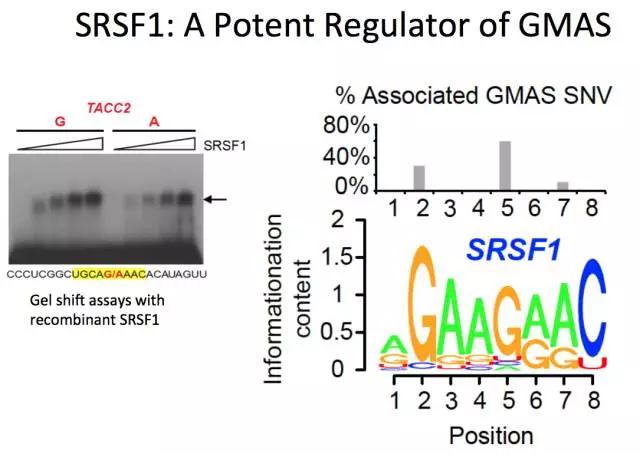

希望这个案例能给诸位课题带来启示。
From 嘉因


